4蛋白质组学
蛋白质组学概述
概念
蛋白质组:一个基因组、一种生物或一种细胞/组织所表达的全套蛋白质
蛋白质组学是研究蛋白质组或应用大规模蛋白质分离和识别技术研究蛋白质组的一门学科,是对基因组所表达的整套蛋白质的分析。
蛋白质组与基因组区别
多样性-同一性;无限性-有限性;动态-静态;空间性-周期性;相互作用-孤立行为;多种技术-单一手段
研究目的和内容
研究目的
了解某种特定的细胞、组织或器官制造的所有蛋白质种类、丰度、功能;
明确各种蛋白质分子是如何形成类似于电路的网络的(相互作用相互关系);
描绘蛋白质的精确三维结构,揭示其结构上的关键部位,如与药物结合并且决定其活性的部位。
研究内容
蛋白质组研究:蛋白质组表达模式;功能模式
蛋白质组研究技术与路线
蛋白质组研究的宗旨--将组织或细胞所有蛋白质(至少是大部分)分离与鉴定
• 蛋白质分离技术
凝胶双向电泳、HPLC(非凝胶电泳);
• 蛋白质鉴定技术
Edman测序、生物质谱技术(当前主流);
• 图像分析与生物信息技术
图像分析软件,数据库;
• 相互作用研究技术
酵母双杂交技术、免疫共沉淀、蛋白质芯片等。
蛋白质分离技术
蛋白质组学研究流程:蛋白质分离、鉴定
蛋白质组凝胶电泳分离技术:SDS-2D-SDS-PAGE
蛋白质组非凝胶分离技术:HPLC、毛细管电泳(CE)
双向凝胶电泳(2D-SDS-PAGE)
二维凝胶电泳又称双向凝胶电泳(two-dimensional gel electrophoresis,2-DE),是目前能将数千种蛋白质同时分离与展示的重要技术。
蛋白质组分析研究的步骤
经典工作流程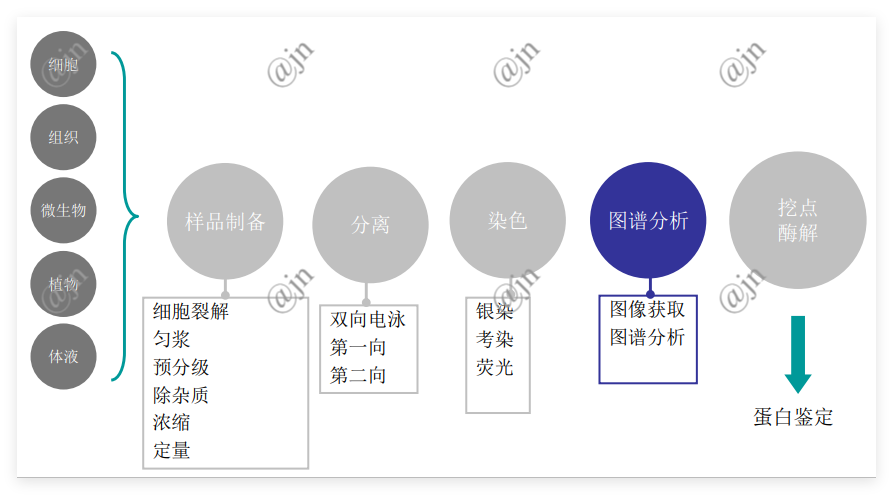
2-DE的原理
两种分离方法的结合:1)根据等电点聚焦(denaturing isoelectric focusing,IEF)分离蛋白质;
2)聚丙烯酰胺凝胶电泳进一步分离聚焦分离的蛋白质。
去污剂SDS-十二烷基硫酸钠,破坏Pr分子的氢键和疏水键,并与之形成Pr-SDS复合物
结果导致:
⑴.复合物带大量负电,不同Pr电荷密度相同
⑵.球状 Pr→雪茄状
用SDS处理后不同蛋白质有着相同的荷质比。在电泳过程中决定迁移率m大小的就是分子筛效应。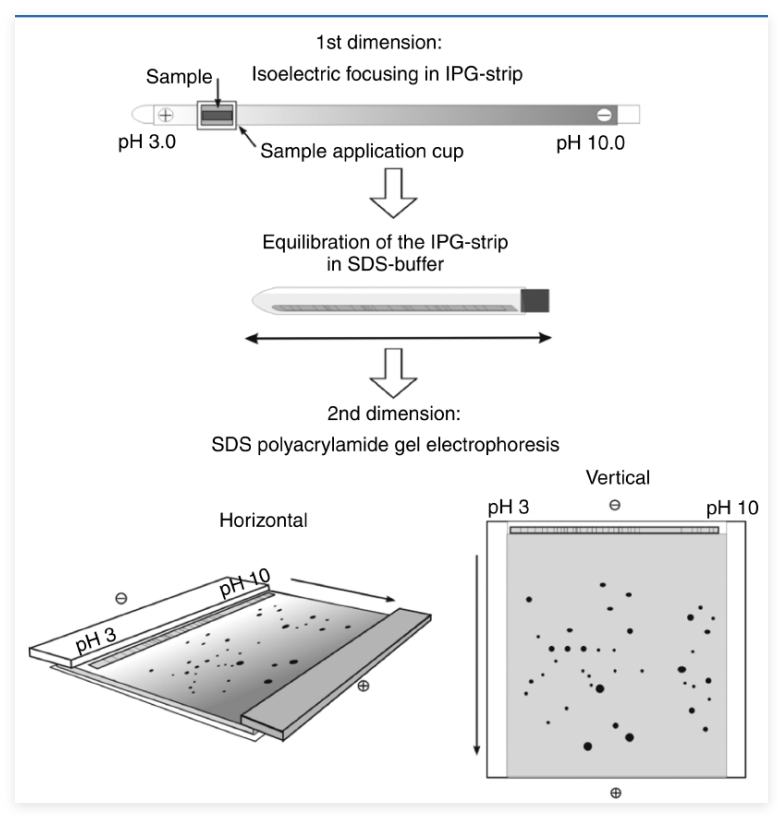
双向电泳样品经过电荷与质量两次分离后,可得到分子的等电点、分子量,分离的结果是点。
胶上蛋白的检测-染色
双向凝胶电泳分离后的蛋白质需要经过染色处理才能进行检测。
目前常用的染色方法:考马斯亮蓝染色、银染、负染、荧光染色、放射自显影
荧光染色
原理
比如用Cy3与Cy5两种染料分别对两个不同蛋白样品进行荧光标记,并在一块2-DE胶上进行运行,由于两种染料激发波长不同,扫描时可得到两张有差异的图象,因此可用于差异蛋白质组学研究。
双向差异凝胶电泳(2D-DIGE)
双向差异凝胶电泳(two dimension difference gel electrophoresis, 2D-DIGE)
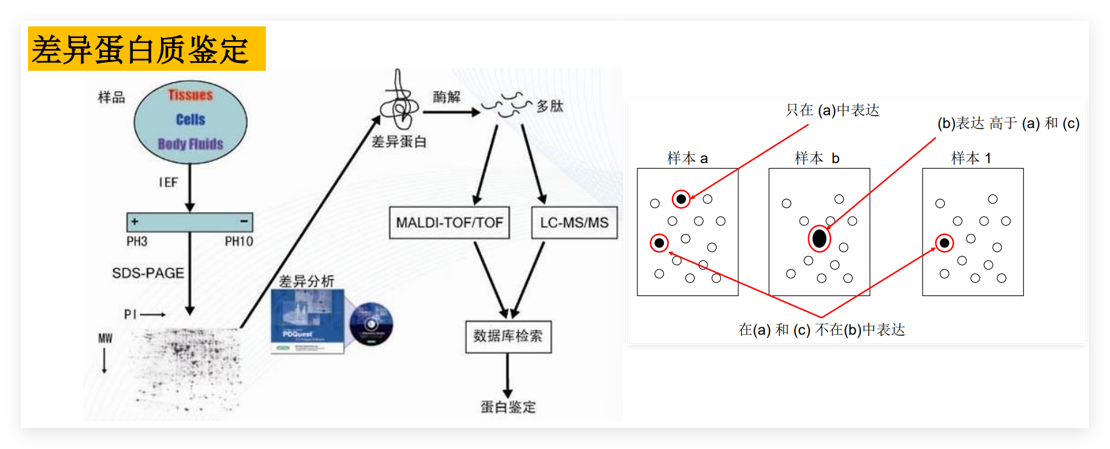
凝胶图谱分析
完成2-DE后,凝胶图像要扫描保存, 以数字化图像的形式储存起来
图像分析内容:
- 蛋白质点数统计;2. 蛋白质点定位;3. 编号;4. 丰度分析;5. 表达差异比较
蛋白质组非凝胶分离技术
高效液相色谱(HPLC)
利用溶质(不同蛋白质)在两相间(固定相和流动相)分配系数、亲合力、吸附能力、离子交换或分子大小不同引起的排阻作用的差别达到分离的目的。
液质联用技术:LC-MS/MS
LC-MS/MS对在双向电泳中难以分离鉴定的高分子量、低分子量、极酸性、极碱性和疏水性强的蛋白进行有效的分离鉴定。
毛细管电泳(CE)
蛋白质的鉴定
主要内容:蛋白质、肽序列测定(一级结构,包含蛋白质的翻译后修饰鉴定)
蛋白质组序列分析方法
(一)Edman降解
蛋白质与异硫氰酸苯酯反应,使N端残基以氨基酸的苯基乙内酰硫脲衍生物的形态游离出来(逐个),然后进行分析(色谱对比)
(二)质谱法(当前主流)
质谱法将蛋白消化成5-25个氨基酸的肽,随即通过LC-MS/MS分析检测,将采集的数据与理论序列数据库进行匹配或者从头测定。
基本原理
应用离子化技术(如高能离子流轰击、化学电离、强电场作用等),使物质分子(气态,或汽化)失去外层价电子形成分子离子,分子离子中化学键进一步发生某些有规律的断裂而形成不同质量的碎片离子(fragment ion)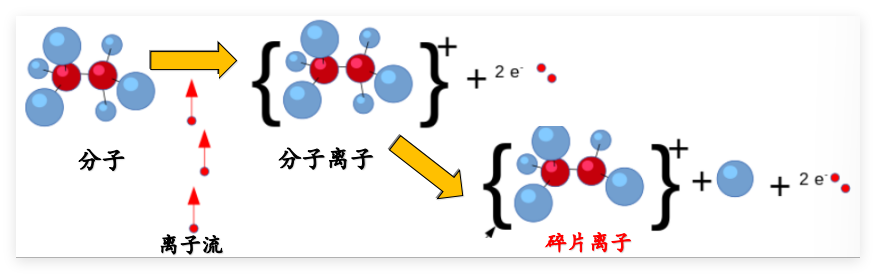
基本原理
带(正)电荷的离子使其在电场或磁场的作用下,根据其质荷比(m/z,离子质量与电荷之比)的差异进行分离,按各离子m/z的顺序及相对强度大小记录的图谱即为质谱
分子离子峰的质荷比就是化合物的相对分子质量,因此用质谱法可测分子量。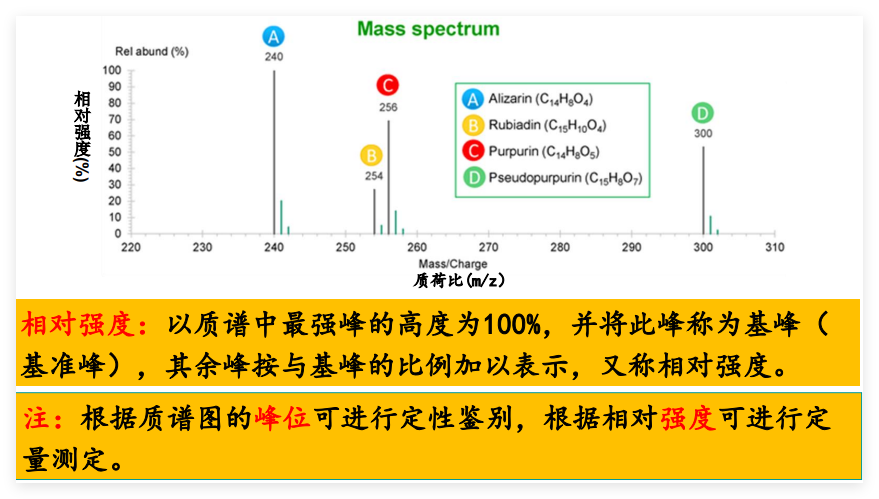
质谱技术介绍
质谱仪组成
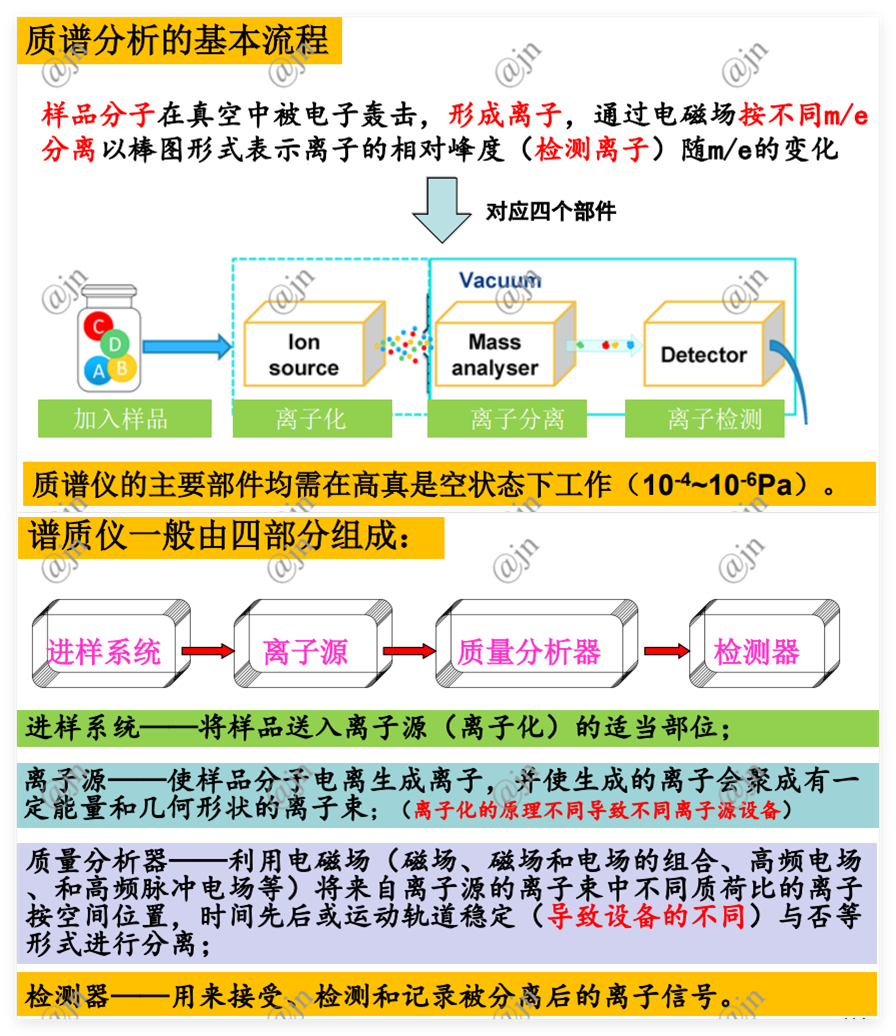
离子源
功能:将进样系统引入的气态样品分子转化成离子。
常用离子源种类:电喷雾电离(ESI);基质辅助激光解析电离(Matrix-Assisted Laser
Desorption Ionization,MALDI)
ESI-MS电喷雾离子化技术
利用强静电场从溶液直接产生气态离子化分子。
进样流出液在高电场下形成带电喷雾。
MALDI-MS基质辅助激光解析电离技术
分析样品置于涂有化学基质的样品板上;基质分子吸收激光能,与样品分子一起蒸发(汽化)并使样品分子电离得到或失去质子。
MALDI技术产生的离子常用飞行时间检测器(TOF)来检测,基质辅助的激光解析飞行时间质谱仪(MALDI-TOF-MS)
质量分析器
将带电离子根据其质荷比加以分离,用于纪录各种离子的质量数和丰度。质量分析器的两个主要技术参数是所能测定的质荷比的范围(质量范围)和分辨率。
- 扇形磁分析器
- 四极杆分析器
- 离子阱分析器
- 飞行时间分析器
有相同动能(离子源提供),不同质量的离子,因其飞行速度不同而分离。如果固定离子飞行距离,则不同质量离子的飞行时间不同,质量小的离子飞行时间短而首先到达检测器。各种离子的飞行时间与质荷比的平方根成正比 - 傅里叶变换分析器
检测器
离子源产生的离子经质量分析器分离的相对强度可以由检测器测量,其结果通常以m/z 值作为横坐标、离子相对丰度为纵坐标的形式表示。
串联质谱
两个或更多的质谱连接在一起,称为串联质谱
联用技术
液相色谱/质谱联用(LC-MS)
色谱可作为质谱的样品导入装置,并对样品进行初步分离纯化,因此色谱/质谱联用技术可对复杂体系进行分离并分析(鉴定)
色谱可得到化合物的保留时间,质谱可给出化合物的分子量和结构信息,故对复杂体系或混合物中化合物的鉴别和测定非常有效。
质谱在蛋白质、多肽分析中的应用
- 分子量的测定
- 蛋白质、多肽纯度的鉴定
- 肽质量指纹谱(重要)
- 肽序列测定技术(重要)
- 蛋白质的翻译后修饰鉴定(了解)
蛋白质组鉴定
肽质量指纹图谱(PMF)
蛋白质的一级结构不同, 其被特定酶解后产生的肽段序列也各异, 形成的肽混合物质量数(分子量)亦具特征性, 称为肽质量指纹谱(peptide mass fingerprint, PMF) , 可用于蛋白质的鉴定。(类似于限制性酶切图片)
PMF流程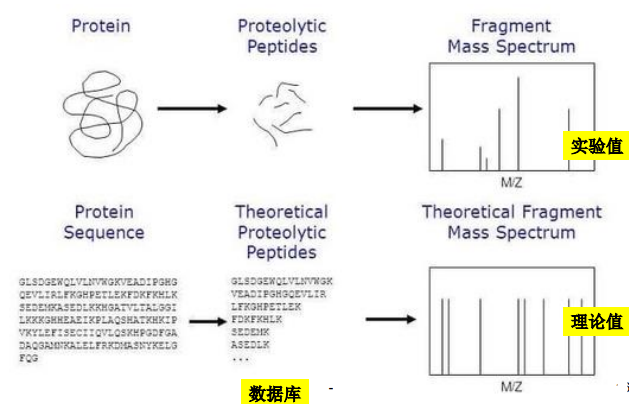
质谱分析蛋白质酶切产生的肽段质量图谱,即PMF,再将PMF中的肽质量离子数据与数据库中理论肽质量离子数据进行比较和评价,从而实现肽段的鉴定
PMF实例:利用MALDI-TOF生物质谱技术结合肽质量指纹谱方法,经过数据库检索,鉴定检测蓖麻毒素(Ricin)
基于串联质谱的肽序列测定
酶水解蛋白质得到的肽段,一级质谱产生肽段前体离子,特定前体离子进入二级质谱。在二级质谱中该前体离子肽骨架酰胺键发生特异性断裂,并产生了一系列可用于确定氨基酸序列的碎片离子,根据质谱图可算出肽序列。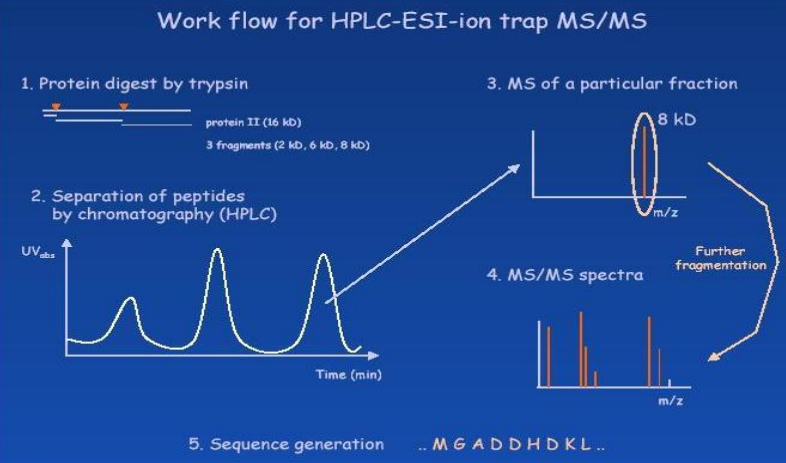
前体离子诱导碎裂会产生的氨基酸片段(四肽为例)
前体离子断裂后产生离子类型
理论上不仅是肽键C-N,还有Cα-C, Cα-N的断裂
保留N末端氨基酸,为 a/b/c型离子;保留C末端氨基酸,为x/y/z型离子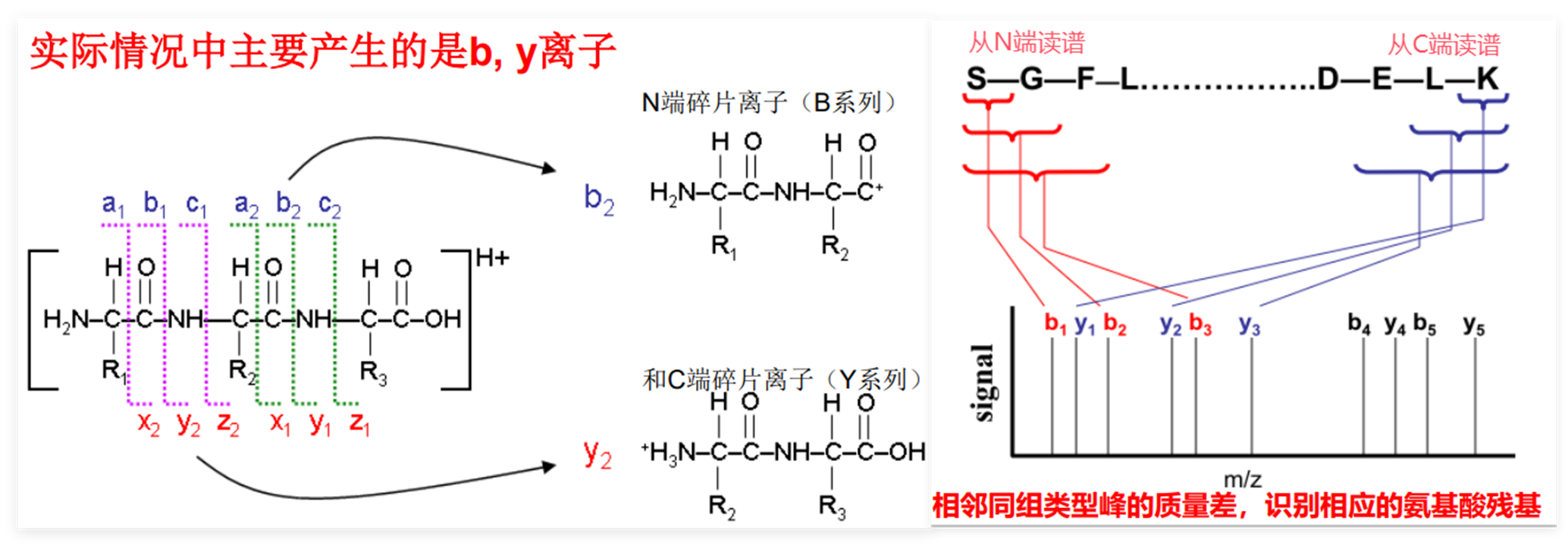
MS/MS测序优点
MS/MS质谱测序可对N 端封闭的肽进行测序(Edman法不可以)
可对修饰的氨基酸残基定性,并确证其位置,包括二硫键的分布
快速、灵敏
定量蛋白质组学技术
常规蛋白质定量分析
确定样品中总蛋白含量,或者某种单一蛋白(纯化后)成分的含量
蛋白质的含量测定
1、基于蛋白质的元素组成特点
2、基于蛋白质的化学显色反应
3、基于蛋白质的光吸收特性
问题:无法实现蛋白质组各个蛋白质(成员)的相对定量分析,根本原因是无法在蛋白质组中区分个体。
基于2-DE的蛋白质组定量分析技术
荧光双向差示凝胶电泳(2D-DIGE,two dimension difference gel electrophoresis)
原理:运用不同的荧光染料(如Cy2, Cy3和Cy5)分别标记不同的蛋白质样品,然后将它们等量混合起来,在单一的双向胶上进行分离和检测,按照它们所发出荧光的不同,来比较在不同细胞状态下蛋白质量的变化。
优点:它克服了在二维电泳过程中不同凝胶间重复性差的问题(引起误差),定量更加准确。
缺点:1. 2-DE不能检测出具极端等电点、分子量太大的、太小的、低丰度的蛋白质及膜蛋白;2. 2-DE分离效果不绝对,许多单一的蛋白质点包含了一个以上的蛋白质。
运用质谱进行定量的蛋白质组分析技术
基本依据:质荷比定性、相对强度定量测定
不同样本分别做质谱分析比较,导致批次误差;不同样本同时做质谱分析比较,无法区分定性
解决方法:增加质荷比差异,让同样的分子具有不同分子量
常见的同位素标记技术
- 化学标记法
(1)ICAT法(同位素亲和标签技术)
(2)iTRAQ法(同重标签标记的相对和绝对定量) - 代谢标记法
(1)15N标记法
(2)细胞培育中含有稳定同位素的氨基酸标记法 (SILAC)
化学标记法
同位素亲和标签技术(ICAT,isotope-coded affinity tags)
ICAT 标签有3部分组成,有两种,重型〔含8个氘〕轻型〔不含氘〕。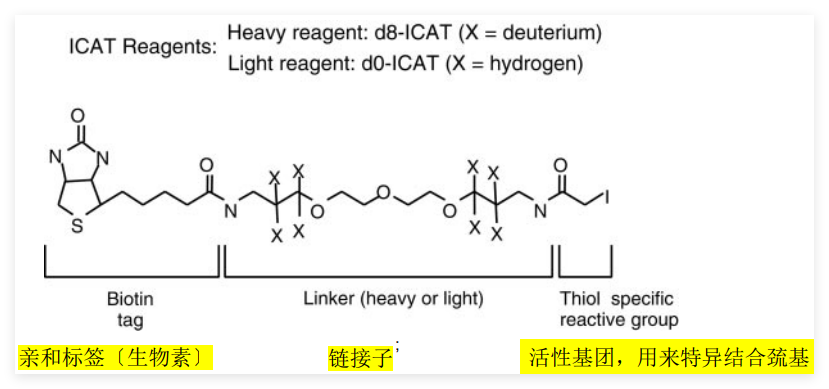
ICAT定量技术流程
① 两种蛋白质组分别用重型ICAT试剂和轻型ICAT试剂标记(巯基反应)
② 标记后等量混合(各个样本总量一样,避免比较基线误差)
③ 胰蛋白酶酶切,得到质谱分析的肽段
④ 亲和纯化得到ICAT标记的多肽(生物素和亲和素)
⑤ 质谱分析,一级MS质谱峰图强度进行定量比较(来自两个样本的肽段成对出现相差 8Da)
⑥ MS/MS进一步断裂差异或感兴趣的肽段,进一步定性确定肽段归属(测序)
一级质谱定量,二级质谱定性。
缺陷
不能用于标记不含半胱氨酸或半胱氨酸含量低的蛋白质
ICAT的相对分子质量约为500 Da,这对肽段来说已经是一个很大的修饰物,会增加数据库搜索算法的复杂性、对一些小的肽段(小于7个氨基酸)更是如此
同重标签标记的相对和绝对定量(iTRAQ)
(isobaric tags for relative and absolute quantitation)
ITRAQ标签构成
报告基因:共有8种,质量数分别为113 ~ 121 Da,最多可同时标记8组样品。
肽反应部分:与肽链N端及赖氨酸侧链发生共价衔接,标记多肽。
平衡基团:质量数为192 ~ 184 Da,使得不同样本的同一肽段具有一样的质荷比。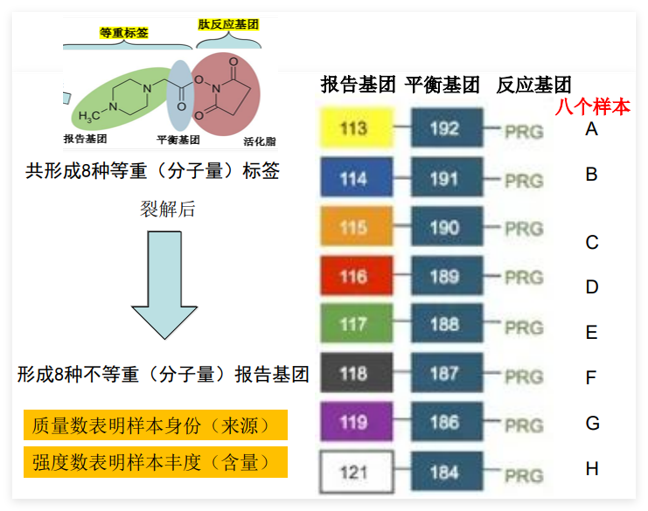
iTRAQ相对定量技术流程
① 对不同的蛋白质样品进行酶切消化成多肽片段
② 使用不同的iTRAQ标签对不同蛋白样品消化产生的多肽片段分别进行标记,但是不同的iTRAQ标签的总质量是一样的。
③ 比例混合各个标记后的样品
④ 带上标记的多肽片段经过一级质谱分离,在一级质谱中,iTRAQ标签由于总质量数一样,来自不同蛋白样品的相同肽段不能被区分。
⑤ 在二级质谱中,在高能碰撞下iTRAQ的平衡基团会被打碎,因此报告基团得到释放。同时肽段也被释放,碰撞碎裂成二级碎片。
⑥ 定性:二级碎片质谱可以推测对应蛋白质的序列;
定量:各个报告基团的在质谱中的信号强度代表多肽在8组样品中的相对丰度,即该蛋白质在8组样品中的相对表达水平
https://www.creative-proteomics.com/resource/understanding-itrack-proteomics-principles-applications.htm
代谢标记法
代谢标记法:样品制备前标记
化学标记法:样品制备后标记
15N标记法
稳定同位素代谢标记技术
其具体作法是,如:在两组酵母细胞的培养基中,一组含有天然氮同位素组分(14N);另一组基质相同,但富含15N。
经过一段时间培养后,将细胞裂解,两组蛋白质等量混合,通过凝胶电泳分离和染色后,分离到特定蛋白质(感兴趣的),将其从凝胶中挖出,酶解后,用MALDI-TOF分析。15N 标记肽的相对分子质量要比14N标记的肽大,通过比较二者在质谱图上的强度,就可以精确定量同一种蛋白质在不同生长条件下的表达量的变化。
SILAC技术
https://www.thermofisher.cn/cn/zh/home/life-science/protein-biology/protein-mass-spectrometry-analysis/protein-quantitation-mass-spectrometry/silac-metabolic-labeling-systems.html
细胞培育中含有稳定同位素标记技术(SILAC,Stable isotope labeling with amino acids in cell culture)
SILAC技术流程
① 标记:在缺乏Lys和Arg的培育基中添加同位素标记的Lys和Arg,培育细胞,使蛋白质被标记成中型或重型氨基酸的蛋白
② 细胞处置,如不同药物处置,或者不同处理时间等等
③ 收集各个实验组的细胞,细胞裂解、提取蛋白质,等量混合。
④ 混合后进行凝胶分离(2-DE,SDS-PAGE),获得感兴趣的蛋白质点
⑤ 割取蛋白质条带,胰蛋白酶消化,质谱分析。
蛋白质的相互作用研究方法
酵母双杂交系统
原理
真核生物转录激活因子
- DNA结合结构域(DNA Binding Domain,BD):与启动子结合,不能激活转录
- 转录激活结构域(Activation Domain,AD):与BD结合,激活转录
组件式特征:结构可互相分开;功能相互独立;空间较近时表现活性;中间序列对活性无影响
被分开的两者通过适当的途径在空间上较为接近时,才能重新呈现完整的转录因子活性,激活特定基因表达。
bait-prey:bait-诱饵蛋白,prey-靶蛋白
酵母双杂交系统组成
酵母细胞:基因组中GAL4基因是缺失的
Gal4为酵母半乳糖苷酶基因的转录激活因子
报告基因:
a.目前应用最多的报告基因是LacZ基因(β-半乳糖苷酶),表达后能在X-Gal (显色底物)培养基上产生蓝色菌落。
b.其次是His基因,表达后能使酵母菌在缺少组氨酸的培养基中生长。(互补生长)
实验设计
(1)为了研究已知蛋白X与未知蛋白Y之间是否能够相互作用,则将GAL4的BD编码区cDNA与蛋白X的cDNA按正确的ORF阅读框连接在一起,构建一重组质粒;同理,将GAL4的AD的cDNA与蛋白Y的cDNA连接在一起,构建另一重组质粒
(2)将两种质粒共转化工程酵母菌(其编码GAL4的基因已缺损),则二者分别表达融合蛋白“BD-X”(“诱饵”,bait)和“AD-Y”)(“猎物”或靶蛋白,prey or target protein)。
(3)若蛋白X和Y二者之间在酵母细胞核内能够发生相互作用,则相当于将原来已拆开的BD和AD又连在了一起,GAL4的活性被恢复,识别并结合于特异的DNA调控元件上,进而激活其下游报告基因(如lacZ)的表达。若X与Y不发生相互作用,则报告基因不表达。即,通过检测报告基因是否表达,就可分析蛋白X与Y二者之间是否发生了相互作用。
应用
a. 验证两种蛋白质之间是否存在相互作用。
b. 筛选已知蛋白质相互作用的新蛋白质。
e. 绘制蛋白质相互作用网络或图谱。
免疫共沉淀技术(Co-IP)
网站:
https://www.antibodies.com/applications/co-immunoprecipitation
定义
免疫沉淀(immunoprecipitation IP)是利用抗体特异性反应纯化富集目的蛋白的一种方法。利用抗体可与抗原特异性结合的特性,将抗原(常为靶蛋白)从混合体系沉淀下来。
原理
在非变性条件下制备总蛋白提取物。以一种蛋白B的抗体
(结合于固相介质)特异地免疫沉淀B蛋白,然后用第二种蛋白或更多种蛋白的抗体做免疫印迹,检测它们是否被第一种蛋白共沉淀,证明相互作用的存在。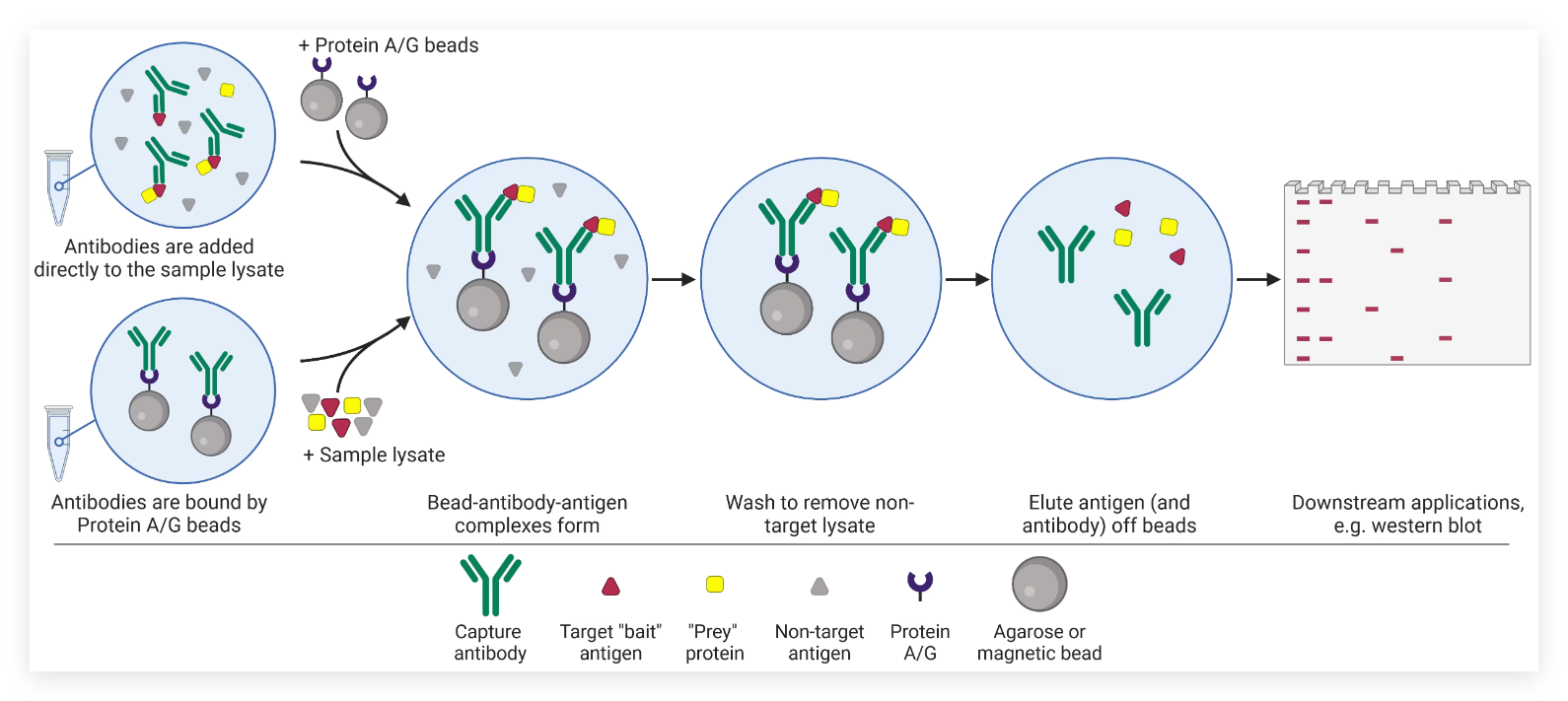
免疫共沉淀总实验流程
(1)蛋白样品准备
(如果为细胞样品,需先裂解细胞)
(2)抗原(目标蛋白)抗体结合反应
(3)Protein A/G 与抗原抗体复合物结合
(4)免疫复合物与 protein A/G 解离
(一般采用 2% SDS 煮沸 5 分钟处理样品)
(5)分析鉴定
(应用 SDS-PAGE,Western-blotting 或质谱仪分析鉴定样品)
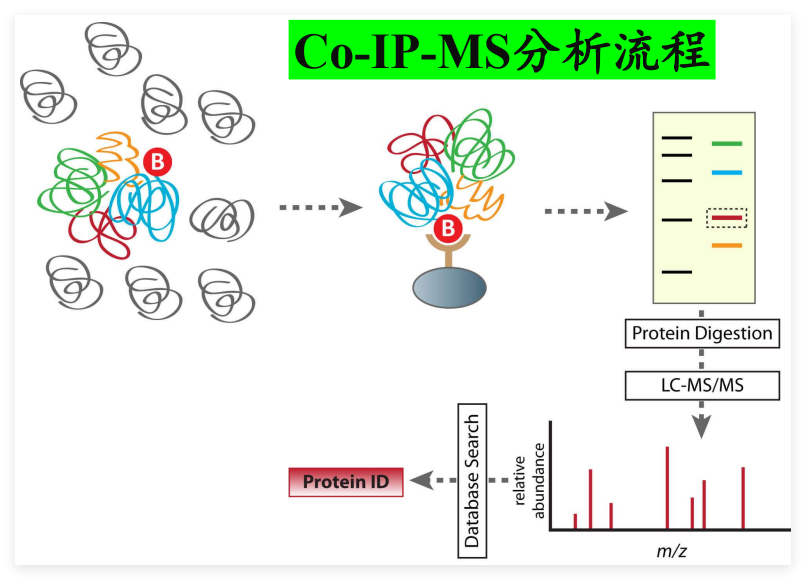
Co-IP与质谱技术联用
原理:以细胞内源性靶蛋白为诱饵,用抗靶蛋白抗体与细胞总蛋白进行免疫共沉淀纯化靶蛋白免疫复合物,凝胶电泳分离后,质谱鉴定靶蛋白的结合蛋白。